Abgeschlossene Projekte
Populationsgenetik und Phylogeographie der Bachforellen in Österreich
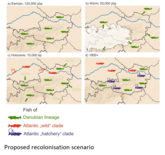
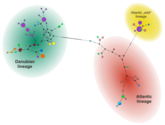
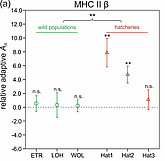
Dieses Projekt zielte darauf ab, die genetische Zusammensetzung von Wild- und Zuchtpopulationen der Bachforellen in Österreich zu beschreiben. Es wurde ein hochauflösendes Typisierungsprotokoll entwickelt, welches 17 Mikrosatellitenloci und die Sequenzierung von >6,000 bp mitochondrieller DNA umfasst. Die untersuchung mehrerer Wildpopulationen ließ uns folgende Hypothese aufstellen: Erst erfolgte eine postglazialen Ausdehnung des Verbreitungsgebietes der sogenannten danubischen genetischen Linie in weite Teile Österreichs. In einer zweiten Welle von Verbreitungsgebiet-Ausdehnungen wanderte auch die atlantische Linie in Gebiete Ober- und Niederösterreichs ein. Im Laufe des 20. Jahrhunderts wurde eine Weitere atlantische Linie durch ausgedehnte Besatzmaßnahmen durch den Menschen eingebracht. Zwischen den rein danubischen Populationen fanden wir starke Unterschiede und Aufspaltungszeiten, welche bis in das letzte Interglazial (vor ca. 120,000 Jahren) zurückdatieren. Während autochthone Wildpopulationen stärker von genetischer Drift beeinflusst zu sein scheinen, beobachteten wir einen relativ stärkeren Selektionsdruck auf die adaptiven Loci in den Zuchtpopulationen. Da die Umweltbedingungen in Zuchten sich meist drastisch von jenen in freier Wildbahn unterscheiden, könnte dieser Selektionsdruck genetische Varianten bevorzugen, welche für Fische, in einer natürlichen Umgebung ausgesetzt, nachteilig sind.
eDNA Untersuchungen zur Fischfauna in der Ötztaler Ache mittels Metabarcoding
Die Fischfauna der Ötztaler Ache wurde mittels eDNA-Metabarcoding erhoben werden. Hierfür werden an 28 Stellen Wasseproben entnommen und die darin enthaltene Umwelt DNA extrahiert. Diese wird mittels Universellen Fisch-Primern amplifiziert und per illumina-Technologie sequenziert.
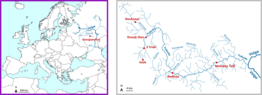
SSqeDNA - Single species targeting via qPCRs in fish monitoring in the headwaters of the Volga river using eDNA
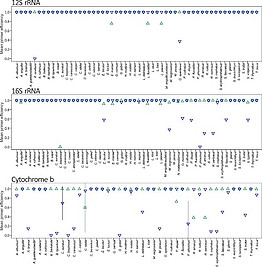
Im Rahmen des Forschungsprojektes “MABeDNA” – Monitoring Aquatic Biodiversity in the headwaters of the Volga River using eDNA” wurde an ausgewählten Standorten des Wolga-Oberlaufs an 11 Probestellen Wasserproben für eDNA Analysen entnommen. Aus diesen Proben wurde die gesamt DNA extrahiert und mittels Metabarcoding die präsente Fischfauna in den jeweiligen Gewässern erhoben. Hierbei konnten erfolgreich 23 heimische aber auch eingeführte Arten dieser Gewässer nachgewiesen werden. Vier Fischarten (Sterlet – Acipenser ruthenus, Aal – Anguilla anguilla, Wels – Silurus glanis und Bachneunauge - Lampetra planeri) konnten mittels Metabarcoding ijedoch in den untersuchten Gewässern nicht nachgewiesen werden. Von diesen Arten gibt es jedoch jeweils an mehreren Beprobungsstellen anderwärtige Nachweise bzw. geeignetes Habitat. In diesem Projekt wurden die Metabarcoding Ergebnisse einerseits dur Erweiterung der genetischen Referenzdatanbanke verfeinert, als auch durch Einzelnachweis-Ansätze (mittesl qPCR) verifiziert. Hierbei konnten Nachweise von vier weiteren Fischarten in diesem System erbracht werden als auch wichtige technische Aspekte zum Metabarcoding Ansatz beleuchtet werden.
Tamara Schenekar
Institut für BiologieUniversitätsplatz 2
8010 Graz